【3D Genome】 Introduction to Hi-C and 3D genomics

This article will introduce the basic concept of Hi-C technology. Hi-C is an analysis method for 3D chromosome organization. With Hi-C, we can observe the interaction between different sites on the chromosome.
何謂Hi-C
Hi-C 全稱為高通量染色體三維構象捕獲技術(High-throughput chromosome conformation capture)利用次世代定序(Next Generation Sequencing, NGS)的快速且便宜的大量定序能力,並結合巨量資料處理以及統計的一種研表觀遺傳學(epigenetics)的利器。主要能建構染色體在三維空間中的互作關係,並搭配其他組學分析(例如ATAC-Seq , ChIP-Seq 等)來觀察染色體相互作用甚至到特定基因位點的調控狀態。
染色質環(Chromatin Loops)被認為是一段基因進行轉錄時需要建立的物理結構,染色質會被塑型元件(例如CTCF)凹成環型,而這微觀的物理結構能讓原本在序列上空間不鄰近的位點(有可能是外顯子,但也可能是增強子或是啟動子等等),轉變成彼此鄰近的位置(這道理有點類似時空扭曲改變物理直線距離的概念),並可能利用這個物理結構來進行各種轉錄工作。染色質環以及相關的結構可以透過Hi-C技術,以資料統計的方式得出。
Hi-C的實驗流程
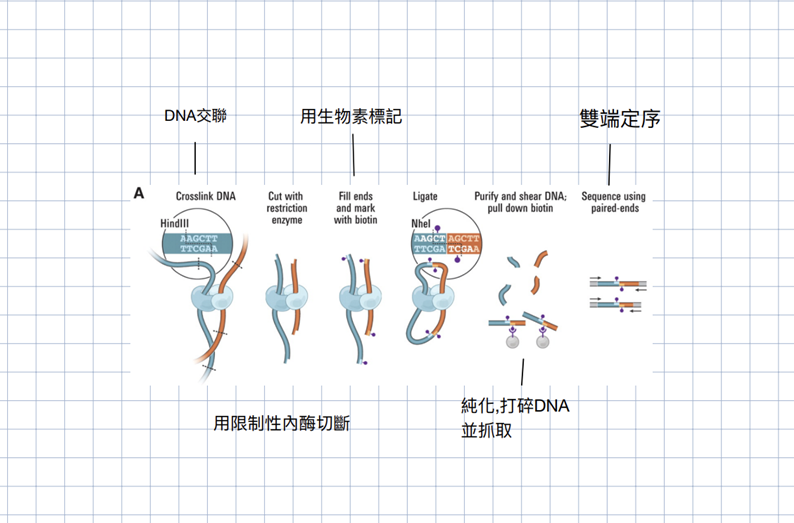
Boxer, L.D. (2010). Hi-C: Genome-wide Chromosome Conformation Capture.Figure2
為了建立任意染色質之間任意位點間的交互作用頻率矩陣,需要先將空間上鄰近的染色質作crosslinking,用限制性內切酶切斷染色質,再將這些片段打碎用後並定序。
定序後的每筆資料會分別來自兩個不同的位置,須將這一對的兩段染色體密碼分別進行比對,並記錄它們分別是來自哪個染色體的位點。再透過數量龐大的成對的資料進行統計,最終就可以建立所有染色體的任意位)點對所有染色體的任意位點之間的關聯頻率,形成一個巨大的矩陣,這裡舉個例如下圖
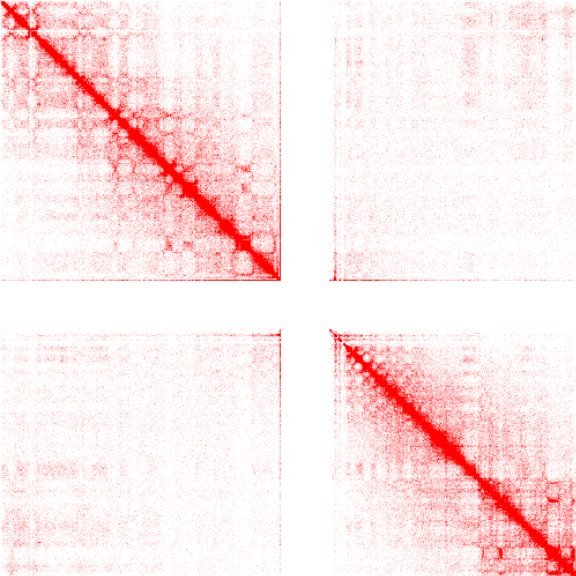
Rao, S. S. P.,et.al. (2017). Cohesin Loss Eliminates All Loop Domains. Cell, 171(2), 305–320.e24. https://doi.org/10.1016/j.cell.2017.09.026
如再搭配基因組學技術,例如經典的ChIP-Seq (染色質免疫共沉澱定序)等方法,就可以建立追蹤從染色體空間互作到關聯的蛋白質或是轉錄因子的運作情況。
Hi-C實務上的難處
Hi-C以及相關的三維度基因組學的難處是定序昂貴到計算的硬體設施對記憶體和硬碟的要求巨大而且昂貴,一個replicate整套處理下來動輒消耗掉幾百GB到TB的儲存空間。(通常在跑定序深度較深的樣本之前,會跑定序較淺的來做測試,這裡指的是定序較深的正式樣本)不過,也是代表進入Hi-C技術的門檻,其中一項是就是同時熟悉HPC環境和Bioinformatics。
Hi-C體驗
Aiden Lab 有開發了一套線上版的Hi-C視覺化工具(https://aidenlab),方便我們˙瀏覽過往Hi-C論文的HiC數據,這邊就直接嵌進來讓大家體驗一下操作:
以上就是HiC技術的簡單分享。
A passionate bioinformatician focuses on the next generation of medical science and biotechnology.



This website was… how do I say it? Relevant!! Finally
I’ve found something which helped me. Appreciate
it!
Thank you! 3D genome organization is a frontier area domain that still need discover. That’s why it’s so charming.