【NGS 次世代基因體資料科學】Gene2vec基因的分散式表徵

這裡紀錄如何利用論文Gene2vec提供的程式,並搭配自己的資料重新訓練模型,訓練基因的分散式表徵。
安裝流程
首先,我們需要建立下載程式的路徑和conda環境,這裡建立python 3.7的虛擬環境:
Bash
mkdir gene2vec_test
cd gene2vec_test/
conda create -n gene2vec_test_env python=3.7
conda activate gene2vec_test_env接著下載程式:
Bash
git clone https://github.com/jingcheng-du/Gene2vec.git
cd Gene2vec/由於gensim 3.x和4在參數名稱上有差異,因此若用舊版需記得去requirements.txt改一下:把gensim>=3.4.0 改成 gensim==3.4.0。(如果希望用新版的,則是需要修改 gene2vec.py ,例如把word2vec的size改成vector_size之類的)
Bash
pip install -r requirements.txt完成之後測試一下:
Bash
cd src/
python gene2vec.py
如果有出現以下訊息說明安裝成功。
Bash
usage: gene2vec.py [-h] N [N ...]訓練模型
接著可以用作者提供的data資料夾作測試,在終端機輸入:
Bash
python gene2vec.py ../data/ ../out txt就會在執行的路徑下輸出gene vector:
Bash
head -n 2 outgene2vec_dim_100_iter_1.txt
FGF6 0.002153057 0.001094015 0.0040485994 0.003507802 -0.0034948308 -0.0032273065 0.002758114 -0.0044576144 0.002355916 0.0017780543 0.004745546 0.0018376901 -0.0035088449 -0.0005739574 -0.000108827386 0.002103943 -0.0038852852 0.0012951874 -0.0031769034 -0.004375249 0.004074314 -0.0026881285 0.004214152 -0.004282877 0.0022233215 0.004169825 0.00061325595 2.0139367e-05 -0.0016913096 0.0025811284 0.0031880501 -0.0019990925 -0.0047910786 0.002188197 0.0026727102 -0.0006805879 0.00019095051 0.0010278132 0.0017754859 0.0031797176 -0.003708027 -0.0043337652 0.0035265626 -0.0008643125 0.00084504695 -0.00054039893 0.0003750502 -0.0037928058 -0.0042927195 -0.0047074244 -0.0017722481 0.00025958134 -0.0026379086 0.00018871028 -0.0019723917 -0.00021585514 0.0033635853 0.0022829815 -0.0024485104 0.0011425553 0.003241704 0.0047381823 0.0012685822 0.0041412427 0.0019761408 0.0019880526 0.0039201365 0.0013327249 -0.002263571 -0.0044547706 0.0037608626 0.00095062394 -0.00030630908 0.0031630904 -0.0018972668 0.004344254 0.0025073248 0.0037321039 -0.004189576 0.0025266777 0.0005846647 0.0019490473 0.0018105969 -0.004199487 0.0020253006 -0.0017606984 -0.004815944 0.0046018823 0.0042982115 0.00051282457 -0.0009345786 0.003392324 -0.0032844574 0.0011845101 -0.0011895953 -0.0012602699 -0.00042309787 0.004582391 0.0025786795 -0.0024350516
GFI1B -0.0029350498 0.0043180487 -0.004318311 0.0019120751 -0.0038370104 -0.00023128637 -0.004420749 -0.0035758333 -0.0040116534 0.0012707855 -0.0009630754 0.0004477923 0.0020208724 -0.00041648198 0.003939566 -0.0040858993 -0.004756729 0.0018472039 -0.0021072265 0.002428173 -0.00014559152 0.0045682737 -0.0033070655 -0.0035072211 0.00053472363 -0.0026147643 0.00052187295 0.0034156216 -0.0035089792 0.001963524 -0.0040159533 0.0029510746 0.004897053 0.0017880275 0.0009832341 -0.004501591 -0.0021778357 0.002407189 0.000616764 -0.003227798 -0.0042902012 0.0024847183 0.003374102 0.002082069 0.001478934 0.0048288074 0.0042617135 -0.0018422379 0.0039390987 0.00026498176 -0.00028904268 0.0011463418 0.0027650178 0.0037835115 0.0007013022 0.004905474 0.0006962089 0.0002940799 0.0038201583 -0.0031658853 -0.00292867 -0.00054527074 0.004884007 0.002188833 0.00015647558 -0.002252723 0.0020673836 0.0038181976 0.00041569016 -0.003276892 -0.002797324 0.0020927635 0.0010414731 -0.004298761 0.002510277 -0.0017390802 0.00439754 -0.0042876415 -0.00071369467 0.002830168 -0.0037963414 0.0036242604 0.00023945107 0.004529737 0.001412234 -0.0010020512 0.0044706156 0.0015063612 0.0029264004 -0.0043842485 -0.0016326424 0.0022118944 0.00042738195 -0.004558031 -0.003733534 -0.0029223813 0.0048098615 -0.0019367941 0.00491898 -0.0025868856 另外可以用已經訓練好的模型,再跑tSNE,以及視覺化,例如:
Bash
pip install MulticoreTSNE
pip install scikit-learn
python tsne_multi_core.py以及python plot.py
Python
import pandas as pd
import matplotlib.pyplot as plt
plt.style.use('ggplot')
df=pd.read_csv("TSNE_data_gene2vec.txt_100.txt",sep=" ",header=None)
df.columns = ['x', 'y']
plt.scatter(x=df["x"],y=df["y"])
plt.show()可以得到gene2vec利用tSNE的2維投影圖:
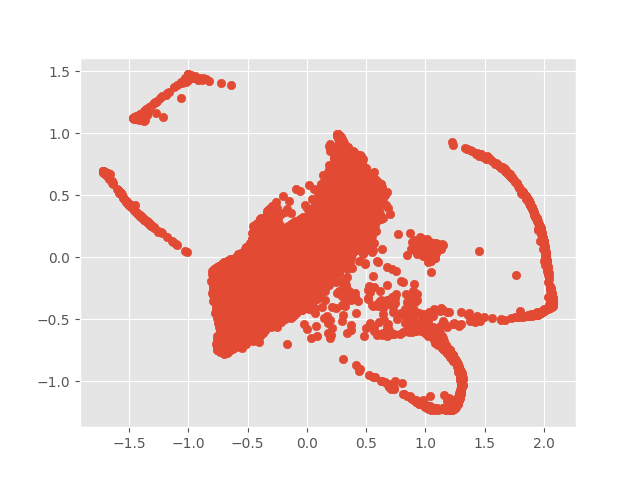
每一個點都是一個gene,這樣我們就大致上完成gene名稱的分散式表徵轉換了。實務上可以將此技術搭配其他marker作預測的應用,來增強預測能力。
而直接去看gene2vec.py的程式可以知道,訓練的方式基本上跟一般NLP詞向量的作法類似,主要是將一般NLP輸入改成GSEA的資料集的基因名單作為輸入。
參考資料
- Du, J., Jia, P., Dai, Y. et al. Gene2vec: distributed representation of genes based on co-expression. BMC Genomics 20 (Suppl 1), 82 (2019). https://doi.org/10.1186/s12864-018-5370-x
是一個對 “下一個世代” 的醫療科技充滿熱血的 Bioinformatican


